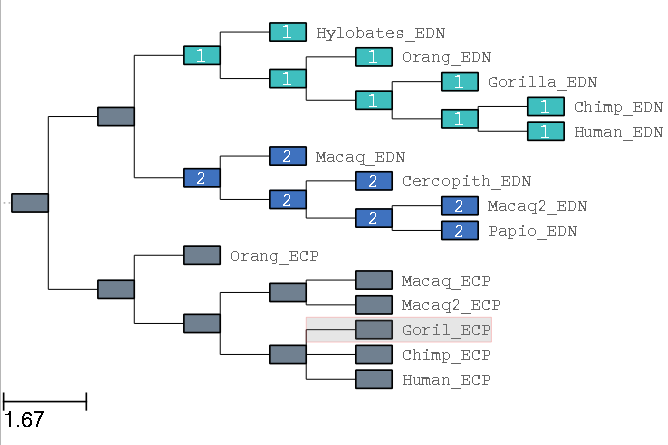
ETE Toolkit — ete-evol による配列解析
ETE Toolkit は系統樹データを扱うための Python フレームワーク。
その中の ete-evol は CODEML や SLR の実行を手助けする Python 製コマンドラインツール。
パッケージとして import して Python スクリプトの中で使うこともできる。
Installation
codeml と slr がインストールされて PATH が通っている必要がある。 (使わない方は入っていなくても解析はできる。)
ete4
# pip3 install lap # if needed
pip3 install https://github.com/etetoolkit/ete/archive/ete4.zipete3
ete-evol は外部ツールに依存するため、 conda を使ってインストールすることが推奨されている:
conda create -n ete3 python=3
conda activate ete3
conda install -c etetoolkit ete3 ete_toolchain
ete3 build check
conda activate ete3conda を介さないインストール:
pip3 install cython bottle brotli numpy scipy PyQt5
pip3 install ete3
ete3 upgrade-external-tools # codeml, slrをここからインストールすることもできる遺伝研
ls /usr/local/biotools/e/ete*2023/09/11時点で ete2 と ete3 が利用可能。 でもちゃんと動かなそう? (バージョンによるかも。)
基本的な使い方
ete3 evol -t tree_file --alg fasta_file -o outdir/ --models models --cpu N-t- 系統樹ファイルのパス
--alg- アライメントファイルのパス
-o- 出力先ディレクトリ。この中にモデルの名前でサブディレクトリが作られる。
--cpu- コア数
--models-
Model Description type Citation M0 negative-selection null Yang 2000 M1 relaxation site 〃 M2 positive-selection 〃 〃 M3 discrete 〃 〃 M4 frequencies 〃 〃 M5 gamma 〃 〃 M6 2 gamma 〃 〃 M7 relaxation 〃 〃 M8 positive-selection 〃 〃 M8a relaxation 〃 〃 M9 beta and gamma 〃 〃 M10 beta and gamma + 1 〃 〃 M11 beta and normal > 1 〃 〃 M12 0 and 2 normal > 2 〃 〃 M13 3 normal > 0 〃 〃 SLR positive/negative selection 〃 Massingham 2005 bsA positive-selection branch-site Zhang 2005 bsA1 relaxation 〃 〃 bsB positive-selection 〃 Yang 2002 bsC different-ratios 〃 〃 bsD different-ratios 〃 Yang 2002, Bielawski 2004 b_free positive-selection branch Yang 2002 b_neut relaxation 〃 〃 fb free-ratios 〃 〃 fb_anc free-ratios 〃 〃
Marking trees
--mark で指定する。スペースで繋ぐことで複数の指定もできる。 2種をカンマで繋ぐ。カンマ3つだと共通祖先からの全ての枝を、カンマ2つだと共通祖先の枝のみ。
Testing evolutionary models
対立仮説と帰無仮説のモデルを --test に渡すことで仮説検定ができる。 例えば M2 vs M1 でサイトモデルの検定をするには、
ete3 evol -t tree_file --alg fasta_file --models M2 M1 --tests M2,M1 -o outdir/のように書く。
対数尤度や p-value は標準出力されるので、 これらが欲しければリダイレクトして取っておく必要がある? ( > output.txt )
モデルの組み合わせ
| 対立仮説 | 帰無仮説 | 検定すること | 引用 |
|---|---|---|---|
| M2 | M1 | 特定サイトにおける正の自然選択 | Yang 2000 |
| M3 | M0 | サイト間でdN/dSが異なるか | 〃 |
| M8 | M7 | 特定サイトにおける正の自然選択 | Yang 2000 |
| M8 | M8a | 特定サイトにおける選択の緩和 | 〃 |
| bsA | bsA1 | 特定の枝の特定のサイトにおける正の自然選択 | Zhang 2005 |
| bsA | M1 | 特定の枝の特定のサイトにおける選択の緩和 | Zhang 2005 |
| bsC | M1 | 特定のクレードの特定のサイトでdN/dSが異なるか | Yang 2002 |
| bsD | M3 | 特定のクレードの特定のサイトでdN/dSが異なるか | Yang 2002, Bielawski 2004 |
| b_free | b_neut | 特定の枝でdN/dSが1と異なるか | Yang 2002 |
| b_free | M0 | 特定の枝でdN/dSが他と異なるか | Yang 2002 |
b_free vs b_neut の検定は P 値とフォアグラウンド枝の \omega = d_\text{N} / d_\text{S} の値によって複数の解釈がある。 (ここについてはドキュメントの記載が間違っている気もする。)
P > 0.05の場合、Relaxed selection の可能性がある。 統計的に主張するにはb_neutを対立仮説にして帰無仮説b_freeを棄却する必要あり?P < 0.05, ω < 1の場合、フォアグラウンド枝でのより強い純化選択の可能性がある。 統計的に主張するにはb_freevsM0で \omega の比較が必要?P < 0.05, ω > 1の場合、正の自然選択を主張できる。