flowchart TD A(Sample) -.->|DNA 抽出| B(DNA) B -.->|NGS| C[Fastq] X[(NCBI)] -.->|prefetch| C C -.->|リードマッピング| D[SAM/BAM] D -.->|バリアントコール| E[VCF/BCF]
全ゲノム解析ハンズオン 2024 新村グループ
スモールデータで理解する SNP 解析の流れ
今日の目標と実施内容
目標
- 公開データを用いた SNP 解析ができるようになる。
- データの中身と解析の流れについて理解を深める。
コンテンツ
- 基本的なコマンドライン操作
- NGS 公開データの取得
- クオリティコントロール
- リードマッピング
- バリアントコール
- SNP アノテーション
Part. 1 コマンドライン操作、データ取得、QC
目標
- 公開データを用いた SNP 解析ができるようになる。
- データの中身と解析の流れについて理解を深める。
コンテンツ
- 基本的なコマンドライン操作
- NGS 公開データの取得
- クオリティコントロール
- リードマッピング
- バリアントコール
- SNP アノテーション
基本的なコマンドライン操作①
cd(change directory)- ディレクトリ📁を移る
mkdir(make directory)- 新しいディレクトリを作成する
ls(list segments)- ディレクトリ内のファイル、ディレクトリを表示する
mv(move)- ファイル、ディレクトリを移動する/名前を変更する
rm(remove)- ファイル、ディレクトリを削除する
基本的なコマンドライン操作②
pwd- 今いるディレクトリのフルパスを表示する
>(リダイレクト)- コマンドの結果をターミナルに表示する代わりに、ファイルに書き込む
head -n N- ファイルの先頭 N 行を表示する (デフォルトは10行)
tail -n N- ファイルの末尾 N 行を表示する (デフォルトは10行)
less-
ファイルを閲覧モードで開く。
Qで閉じる。
基本的なコマンドライン操作の練習🔰
- 遺伝研スパコンにログインし、今日の実習用のディレクトリ
snp24を作成する。 snp24内に移動し、今いるディレクトリのフルパスをターミナルに出力する。- 今いるディレクトリのフルパスを、ファイル
fullpath.txtに書き込む。 fullpath.txtのファイル名を、pwd.txtに書き換える。pwd.txtの中身を確認する。pwd.txtを削除する。
解答例
mkdir snp24cd snp24してpwdpwd > fullpath.txtmv fullpath.txt pwd.txthead pwd.txtとか、less pwd.txtとか。rm pwd.txt
今日のハンズオンはすべて snp24 ディレクトリの中で行います。
ハンズオン終了後は、ディレクトリごと消してもらって構いません:
SNP 解析の流れ
- リードマッピング
- リードを参照配列の相同な位置に貼り付ける
-
![]()
- バリアントコール
- 参照配列と異なる配列を特定する
-
![]()
今日使うデータの説明
大腸菌 Escherichia coli (E. coli)
- ゲノムサイズが約 4.6Mb と小さい
(ニワトリで約 1Gb) - 参照配列: B str. REL606 (Ensembl47)
- SRA ショートリード: SRR030257
- なんのデータ?
- 高温条件下での進化実験 20,000世代 (Barrick et al. 2009)
- 問い
- 高温条件で進化させるとどんな遺伝子に変異が入るのか?
今日使うソフトウェア一覧
公開データの取得
研究で読まれた配列データは NCBI などのデータベースにアーカイブされ、だれでも利用できる。
flowchart LR A(Sample) -.->|DNA/RNA 抽出| B(DNA/RNA) B -.->|アーカイブ| C[(NCBI)]
どうやって探すか:
- NCBI で検索する
- 論文の Data availability などから BioProject ID を探す
(例: Bendesky et al. 2024)
今回使うデータをみてみる
- NCBI にアクセスして、 SRR030257 を検索
- Genomes タブの SRA をクリック
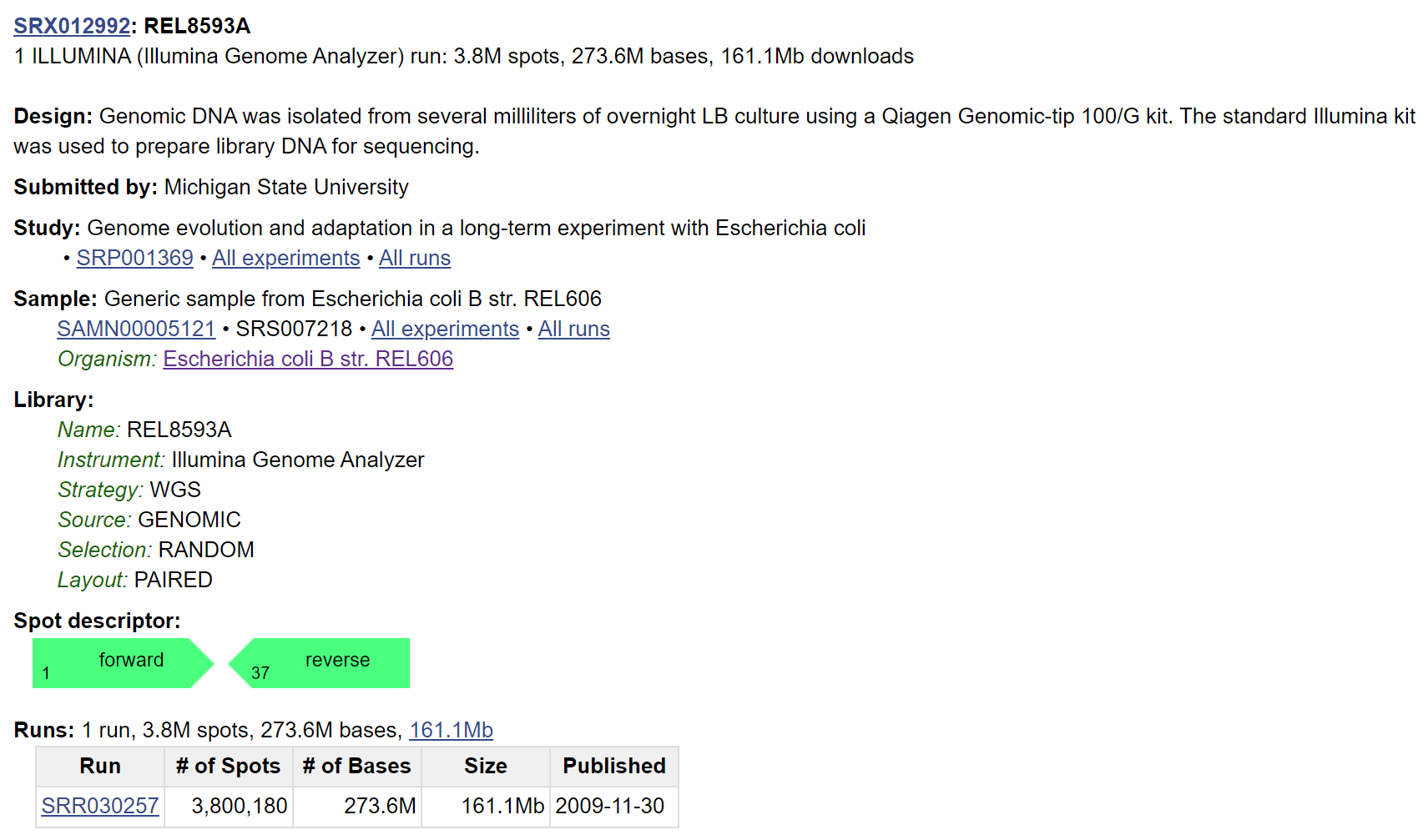
← ダウンロードにはこの Run ID が必要
SRA Toolkit を使って配列をダウンロードする
https://github.com/ncbi/sra-tools/wiki/08.-prefetch-and-fasterq-dump
prefetch- SRA アクセッションをダウンロードする
fasterq-dump- SRA アクセッションから Fastq ファイルを取り出す
ls して2つの .fastq ファイルができていたら成功:
Fastq ファイルの中身を見てみる
4行でひとまとまり (1リード) のデータ:
クオリティ値
Fastq の4行目は、各塩基のクオリティ値が記号で記されている。
記号と数字の対応は以下の通り (ASCII コード):
これらの数字 (Q値) は Log スケールであり、ある塩基が間違って読まれている確率を \(P\) として、
\[ Q = -10 \times \log_{10}{P} \]
で計算される。
例えば ? なら \(Q=30\) で、間違って読まれている確率は \(P=0.001\)。
NGS 配列のクオリティコントロール
- 低品質な (Q値の低い) 塩基
- アダプタ配列の混入
- PCR duplicates
- 他サンプルのコンタミネーション
- など
偽陽性バリアントの増加
クオリティコントロールのツール
| Tool name | language | input | QC | QF | Ad | cont | PE |
|---|---|---|---|---|---|---|---|
| FastqPuri | C, R | fq | 〇 | 〇 | 〇 | 〇 | 〇 |
| fastp | C++ | fq, gz | 〇 | 〇 | 〇 | x | 〇 |
| Fastq Screen | perl | fq | x | x | x | 〇 | x |
| trimmomatic | java | fq, gz | x | 〇 | 〇 | x | 〇 |
| FastQC | java | fq, gz | 〇 | x | x | x | x |
| RSeQC | C, Python | BAM/SAM | 〇 | x | x | x | x |
Pérez-Rubio et al. 2019 Table. 1 より抜粋
QC: Quality Control
QF: Quality Filtering
Ad: アダプタ配列除去
cont: コンタミ除去
PE: Paired-end 対応
今回のハンズオンでは、C++ 製で高速、
圧縮ファイル (gz) にも対応した fastp を使う。
fastp によるクオリティコントロール
-i,-I-
入力 Fastq ファイル。Single-end の場合
-iのみ。 -o,-O-
出力 Fastq ファイル。Single-end の場合
-oのみ。 -
.gzをつければそのまま圧縮可能。 -h-
レポートファイル (
.html) の出力先
fastp によるクオリティコントロール
-q- クオリティ値の下限 (デフォルトは15)
-u- 基準を下回る塩基が何%以上含まれているリードを除くか (デフォルトは40)
今回の場合、\(Q<20\) の塩基が40%以上含まれれるリードを除去。
Part. 1 まとめ
達成🎉
参考

