Minimal hands-on for Comparative Genomics
ダイジェスト版 バイオインフォマティクス
生物多様性の源は?
現在の生物の多様性・複雑性は、単一の共通祖先に端を発する進化の歴史の中で形成されてきた。

 https://en.wikipedia.org/wiki/
https://en.wikipedia.org/wiki/
生物の「進化」とはなにか
生物の性質が、世代を経るにつれて変化すること。
↓ ちょっとアバウトすぎる
親から子へと受け継がれる情報が変化し、その変化がやがて集団中に広まっていくこと。
↓ もう少し学術的にいうと
親から子へ遺伝する情報に変異が生じ、集団中における頻度が変化すること。
キリンの例: (こんなに単純ではないだろうけど)
- とある遺伝子に変異が生じた結果、首の長い個体が現れた。
- 首の長い個体は高いところの葉っぱも食べることができて生存に有利。
- 首の長い個体ばっか生き残った結果、キリンの首が長くなった。
決して高いところの葉を食べようとして首を長くしたわけではない。

遺伝情報? 遺伝子?
- 遺伝情報(親から子へ伝わる情報)の実体
- DNAの配列 = DNAを構成する4種類の塩基(A, T, G, C)の並び
- 遺伝子
- DNAのうち、タンパク質(体を形作る素材)をつくる領域
 https://theory.labster.com/
https://theory.labster.com/
バイオインフォマティクスとは
生物の持つ膨大な情報(主に遺伝情報)を、コンピュータの力を借りて扱う学問。
- ある生物の遺伝情報(A, T, G, Cの並び)はどうなっているのか
- ある種と別の種の遺伝情報はどこが同じで、どこが違うのか
- 遺伝情報はどう変わってきたのか、どう変わりつつあるのか
本日は野良データ + Python + Linuxで、バイオインフォマティクスの一部を体感。



本日のお品書き
野良データを使って、バイオインフォマティクスの解析の一部を体験してもらう。
ペットとしてお馴染みのイヌ( Canis lupus familiaris )と、オーストラリアに生息するイヌの近縁種であるディンゴ( Canis lupus dingo )のゲノムを、重複遺伝子の観点から比較する。

- CONTENTS -
- 重複遺伝子とは?
- 種の環境適応と重複遺伝子
- 重複遺伝子を調べるゲノミクス手法
- Let’s try on your computer!
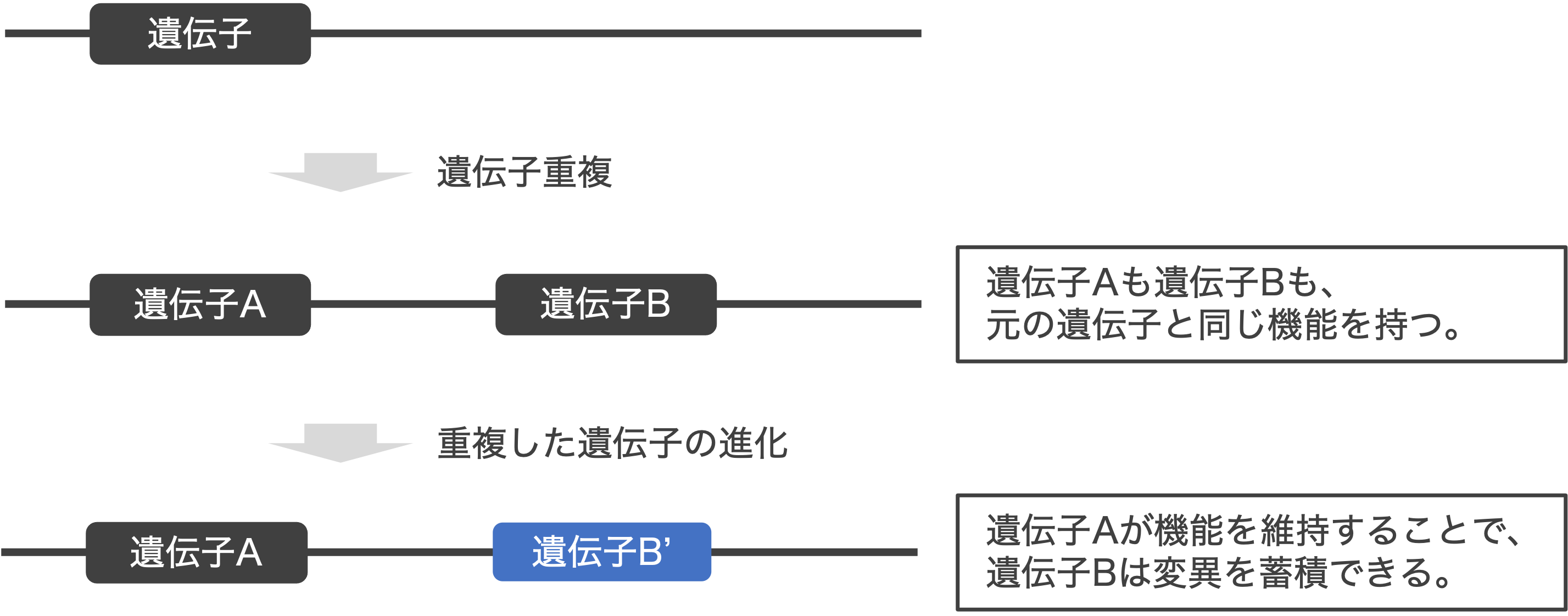
重複遺伝子とは
もともと1つだった遺伝子が、さまざまな要因(組換えのミス、トランスポゾンの転移、etc.)で2つ以上になること。
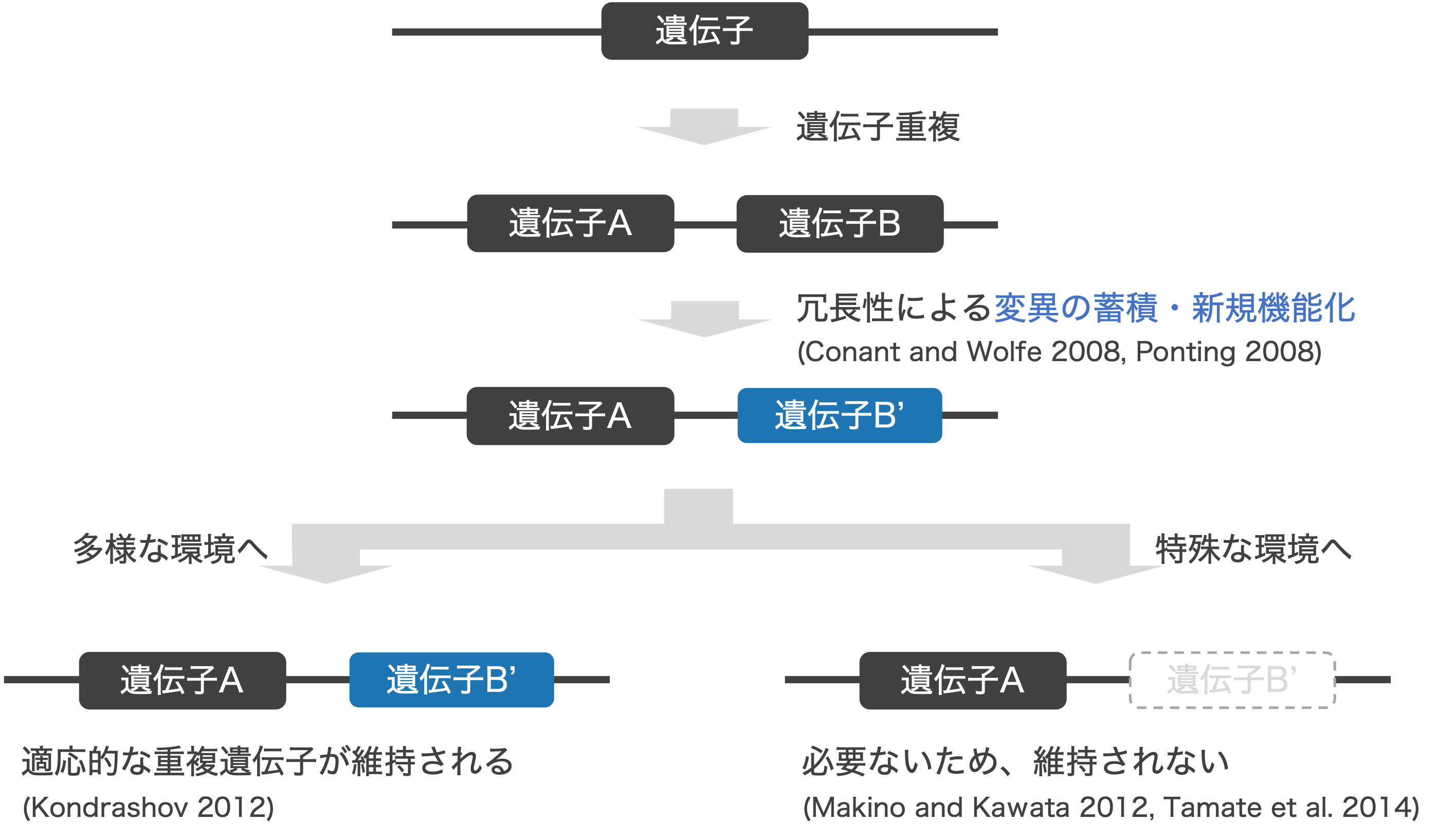
重複遺伝子は環境への適応に関与する
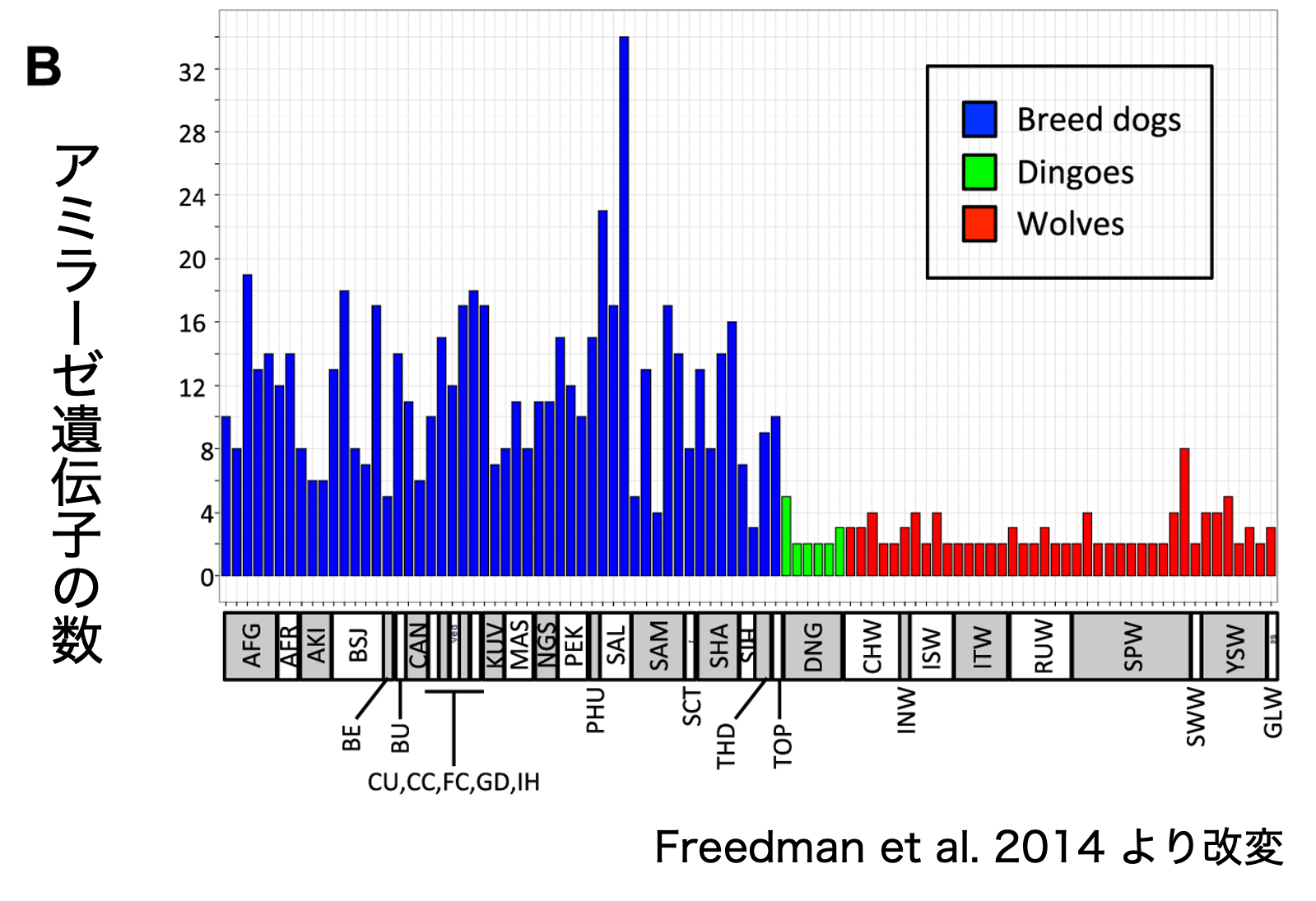
アミラーゼ遺伝子の数
- アミラーゼはでんぷんを分解する消化酵素。いろんなものを食べるヒトは5つのアミラーゼ遺伝子をもつ。:
AMY1A,AMY1B,AMY1C,AMY2A,AMY2B

 左: αアミラーゼ 右: βアミラーゼ
左: αアミラーゼ 右: βアミラーゼ
これまでの話と絡めると、多様な食事を摂る環境ではアミラーゼ遺伝子の重複が有利に働いたと言える。
では、長く人間のペットとして生きてきたイヌと、野生に暮らすディンゴでは、どちらの方がアミラーゼ遺伝子を多く持つだろう?
重複遺伝子を調べるためのゲノミクス手法
重複遺伝子どうしはもともとは1つの遺伝子(=同じ配列)なので、互いに似た配列を持つ。そこで、
- BLAST (Basic Local Alignment Search Tool)
- DNAの塩基配列あるいはタンパク質のアミノ酸配列のアライメントを行うためのアルゴリズム
- ある閾値以上のスコアで類似する配列を発見することができる。

実際にやってみよう: 前準備
homebrewのインストール:
blastのインストール:
今回使うリポジトリを取ってくる:
実際にやってみよう:
minicongに移動イヌとディンゴの配列を取得
blastpを実行 (1行ずつやってみてもいいよ)結果を確認してみる。
アミラーゼ遺伝子の数は イヌ > ディンゴ
ヒトと共に生活し始めたことで雑食性に変化したこととの関与が示唆されている。
See also > イヌの多様化における遺伝的背景(かずさDNA研究所)
バイオインフォマティクスは生物学における強力な研究手法
ゲノムを読む技術の発達に伴い、日々多くの種のゲノムが解読されている。バイオインフォマティクスは、蓄積されつつある大規模な情報を扱い多様な生命現象を解き明かす強力なツールである。

- SEE MORE >
- 東北大学 生命科学研究科 進化ゲノミクス分野
